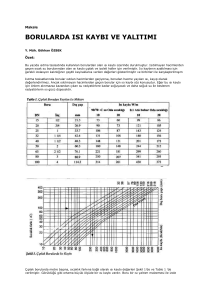
øZMøR KÖRFEZø`NDE GELøùEN BøR KAHVERENGø ALG OLAN
advertisement

øZMøR KÖRFEZø’NDE GELøùEN BøR KAHVERENGø ALG OLAN Cystoseira mediterranea SAUVAGEAU’DAN DNA øZOLASYONU øNCø TÜNEY* ,ATAKAN SUKATAR*, SACøDE PEHLøVAN** *Ege Üniversitesi Fen Fakültesi Biyoloji Bölümü Hidrobiyoloji A.B.D. Bornova/øzmir **Ege Üniversitesi Fen Fakültesi Biyoloji Bölümü Genel Biyoloji A.B.D. Bornova/øzmir ÖZET Deniz alglerinden DNA izole etmek üzere yayınlanmıú çok sayıda yöntem bulunmaktadır. Bu yöntemler genellikle di÷er organizmalar için kullanılan DNA izolasyon yöntemlerden de÷iútirilerek ortaya çıkarılmıútır. Deniz alglerinden DNA izolasyonu, onların talluslarında bulunan polisakkarit ve polifenolik bileúiklerin bollu÷u nedeniyle oldukça zordur. Algler içerisinden özellikle kahverengi alglerde bu zorluk karúımıza çıkmaktadır. Bu çalıúmada, kahverengi alglerden Cystoseira mediterranea Sauvageau’da DNA izole etmek için kullanılan en yaygın yöntemlerden biri olan CTAB (cetiltrimetilamonyum bromid) yöntemi modifiye edilerek uygulanmıú ve moleküler analizlerde kullanılacak kalitede DNA izolasyonu baúarılarak PCR yönteminde kullanılmıútır. Anahtar Sözcükler: DNA izolasyonu, CTAB, Kahverengi Algler THE DNA EXTRACTøON PROTOCOL FROM ONE OF THE BROWN ALGAE Cystoseira mediterranea SAUVAGEAU øN IZMøR BAY ABSTRACT There are various methods published about DNA extraction from marine algae. These methods are the modifications of several DNA extraction methods from other organisms. Extraction of DNA from seaweeds are difficult processes because of the polysaccharide and polyphenole compounds of their thallus. In this study, DNA from a brown algae from (Cystoseira mediterranea Sauvageau ) collected from the Bay of Izmir was extracted by using modified CTAB (cetiltrimethylamonium bromide) protocol and used in PCR analysis. This modified method was also found efficient and applicaple for other molecular purposes. Key words: DNA extraction, CTAB, Brown Algae GøRøù Kahverengi algler, ticari önem taúıyan cinsleri kapsayan önemli bir alg sınıfıdır. Metabolitlerinin alginat eldesinde kullanılması, antimikrobiyal aktiviteye sahip olması, eczacılık ve kozmetik sanayinde, ka÷ıt üretiminde, tekstil sanayi gibi birçok alanda kullanılması ve birçok denizde bulunmaları nedeniyle büyük öneme sahiptirler. Türlerin tanımlanmasında kullanılan en yaygın yöntem morfolojik özelliklere bakılarak tür ayrımının yapılmasıdır. Ancak bazı türlerin benzer morfolojik özellikleri paylaúması ya da aynı türün úekillerinin çevre úartlarına ve büyüme evrelerine göre farklılıklar göstermesi, tür tayinlerinde en çok karúılaúılan problemlerdir. Bu nedenle günümüzde daha duyarlı ve kesin sonuç veren yöntemlere gereksinim duyulmaktadır. Son yıllarda bir çok alanda kullanılan moleküler yöntemler, çeúitli organizmaların tür tanımının yapılmasında ve ayrıca türler arasındaki akrabalık iliúkilerinin ortaya çıkarılmasında da önemli ölçüde kullanılmaktadır. Bir organizmanın tanımlanabilmesi için yapılan moleküler analizlerde öncelikle o örne÷in DNA’sının elde edilmesi gerekmektedir. øzole edilen DNA’nın kalitesi uygulanacak olan analizlerin sonucunu etkiler. Her organizma hatta her cins için farklı DNA izolasyon yöntemleri bulunmakta ve en etkilisinin bulunması için araútırmalar devam etmektedir. Kahverengi alglerden DNA izolasyonu onların talluslarında bulunan polisakkarit ve polifenolik bileúiklerin çok fazla bulunması nedeniyle zor olması ve çok fazla örne÷e gereksinim duyulması gibi nedenlerle, moleküler bazlı evrimsel çalıúmaların yapılmasını zorlaútırmaktadır. Elde edilen DNA’nın kalitesini arttırmak için birçok polisakkarit için uzaklaútırma yöntemi geliútirilmiútir. Bunlar; CsCl ultrasantrifüj yöntemi (Sosa & Oliveira, 1992), Agaroz jel elektroforezi saflaútırması (Saunders, 1993), Hidroksiapatit kolon saflaútırması yöntemi (Dutcher et al.,1930) vb.. Tüm bu yöntemler yüksek kalitede DNA eldesi sa÷lasa da oldukça karmaúık, zaman alıcı ve çok miktarda örne÷e gerek duyulan yöntemlerdir. Bu nedenle populasyon bazlı çalıúmalar için pek uygun de÷illerdir. Bu çalıúmada, beú farklı yöntem de÷iútirilerek ülkemizde geliúen kahverengi alglerden Cystoseira mediterranea Sauvageau örne÷inde denenmiútir. Burada kullanılan DNA izolasyon yöntemleri, farklı alg gruplarına ba÷lı olarak optimize edilmesi gereken çeúitli basamaklardan oluúmaktadır. Bu çalıúma sonucunda C. mediterranea örne÷i için 101 uygun olan iki izolasyon yöntemi belirlenmiú, modifiye edilerek uygulanmıú ve PCR’da kullanılarak izolasyon kalitesi gözden geçirilmiútir. MATERYAL VE YÖNTEM Materyal olarak øzmir Körfezi’nde geliúen, kahverengi bir alg olan C. mediterranea toplanmıútır.Örnekler 8 ùubat 2005 tarihinde Urla Hekimadası ve çevresinden elde edilmiú, toplandıktan sonra saf suda yıkanarak, so÷uk zincirde laboratuvara getirilmiútir. Epifitlerinden ayıklandıktan sonra tekrar saf suda yıkanmıú ve daha sonra -200C’de saklanmıútır. øzolasyon için yöntem baúına 20 mg. örnek kullanılmıútır. CTAB (cetiltrimetil amonyum bromid) çözeltisi: 5M NaCl, 1M Tris-HCl (pH=8), 4g. CTAB, 0.5M EDTA (pH=8) ve distile su kullanılarak hazırlanıp, iyice karıútırılarak otoklavda sterilize edilmiútir. øzolasyonda CTAB çözeltisinin yanı sıra kloroform: izoamil alkol (24:1), TE buffer, Etanol, ȕ-ME (ȕ-Merkaptoetanol) ve PVP (Polivinilpirrolidon) kimyasalları kullanılmıútır. Tüm kimyasallar otoklavda sterilize edildikten sonra kullanılmıútır. øzolasyon için aúa÷ıdaki basamaklar uygulanmıútır: øzolasyona baúlamadan önce CTAB çözeltisi ve ȕ-ME karıútırılarak 650C’de 1 saat ısıtılıp, 20 mg. donmuú doku sıvı nitrojende iyice ö÷ütülüp toz haline getirildi. Ö÷ütülen doku eppendorf tüpüne koyularak üzerine ȕ-ME ve CTAB çözeltisi karıúımı eklenerek, 45 dakika 650C’de inkübasyona bırakıldı Ardından tüpe kloroform: izoamil alkol (24:1) eklenerek 10 dakika proteinlerin çökmesi için çalkalandı ve 5 dakika santrifüj yapıldı . Üstte kalan sıvı faz yeni bir eppendorf tüpüne aktarıldıktan sonra tekrar kloroform: izoamil alkol (24:1) eklenerek 10 dakika çalkalandı ve santrifüjün ardından sıvı faz yeni bir appendorf tüpüne aktarıldı Üzerine so÷utulmuú izopropanol eklendi. 200C’de 30 dakika ile 8 saat arası bir kaç inkübasyon süresi denenerek DNA’nın çökelmesi beklendi. ønkübasyonun ardından 5 dakika santrifüj yapıldı ve üstteki sıvı faz atıldı . Kalan çökeltinin üzerine TE buffer eklenerek 370C’de çözünmeye bırakıldı . Ardından 3M NaAc ve hacim %96 etanol eklenerek oda sıcaklı÷ında çözünmeye bırakıldı. 10 dakika santrifüj edildikten sonra sıvı faz atıldı. Kalan çökelti %70 so÷uk etanol ile yıkanıp 2 dakika santrifüj edildi ve sıvı faz atıldı. Yaklaúık 5 dakika etanol buharlaúana kadar santrifüjde kurutuldu ve TE bufferda 370C’de çözdürülüp DNA +40C’de saklandı. Kullanıma uygun bulunan di÷er DNA izolasyonu yönteminde, ilk basamakta kullanılan CTAB ve ȕ-ME karıúımına PVPP maddesi eklenerek izolasyon yapıldı. Ayrıca birinci yöntemde kullanılan NaAc yerine ikinci yöntemde KAc kullanıldı. Son basamakta ise TE buffer kullanıldıktan sonra 550C’de 1 saat inkübasyona bırakıldı ve daha sonra da +40C’de saklandı. ùekil 1: Sırasıyla 5 farklı yöntem sonucu elde edilen agaroz jel elektroforezi görüntüleri. ùekil 2: II. ve III. yöntemlerin farklı inkübasyon süreleri kullanılarak uygulanmıú úekillerinin agaroz jel elektroforezindeki görünümü. 102 BULGULAR VE TARTIùMA Tablo I. øzolasyon Çözeltisinde Kullanılan Baúlıca Kimyasal Madde Miktarları Bu çalıúma, C. mediterranea türünden, moleküler analizlerde kullanmak için uygun DNA izolasyon yöntemini bulmak amacıyla yapılmıútır. Uygulanan 5 yöntem arasında Stefan Kraan’ın kahverengi algler için CTAB-PVP DNA ekstaksiyon protokolü (I. Yöntem), Doyle & Doyle (1987)’ün ve Cullings (1992)’nin DNA izolasyon yöntemlerinin de÷iútirilmiú úekli (II. Yöntem), Soltis Lab. CTAB DNA ekstraksiyon protokolü (III. Yöntem), Hombergen & Bachman (1995)’nın Gabrielson et al.(1997) tarafından modifiye edilen izolasyon yöntemi (IV. Yöntem) ve Colosi & Schall (1993)’ın DNA izolasyon yöntemleri (V. Yöntem), bulunmaktadır. Yöntem I II III IV V CTAB (ml) 100 40 40 40 40 PVP (g) 0,2 200 - ȕ-ME (µl) 1,5 400 1,6 80 40 Alglerden DNA izole etme yöntemlerini, bitki örneklerinden DNA izole etme yöntemlerinden ayıran temel fark, alglerde izolasyon sırasında PVP ve ȕ-ME kimyasallarının kullanılmasıdır. Bu maddeler özellikle kahverengi alglerde çözeltideki oranları arttırılarak kullanılmaktadır. Yapılan bu çalıúmada beú farklı yöntemde kullanılan CTAB, PVP, ȕME oranları Tablo I’de verilmiútir. øzolasyon sonunda elde edilen DNA miktarına bakıldı÷ında en yüksek kalitede DNA miktarının II., III. ve V. yöntemlerden elde edildi÷i görülmüútür (ùekil I ve II). Bu yöntemlerdeki kimyasalların miktarına bakıldı÷ında sadece III. yöntemde PVP’nin kullanıldı÷ı ve buradaki DNA’nın agaroz jel elektroforezinde yürütüldü÷ünde en parlak DNA bandının bu yöntem ile elde edildi÷i belirlenmiútir. øzolasyon çözeltilerinde içerik olarak bu üç kimyasaldan sadece CTAB ve ȕ-ME bulunan II. ve V. yöntem karúılaútırıldı÷ında, II. yöntemle elde edilen DNA bantlarının V. yöntem ile elde edilen DNA bantlarından daha parlak oldu÷u saptanmıútır. Bunun sebebinin kullanılan ȕ-ME miktarının daha fazla olmasından kaynaklandı÷ı düúünülebilir. ølk analizler sonucunda en verimli üç yöntemin, Doyle & Doyle (1987)’ün ve Cullings (1992)’nin DNA izolasyon yöntemlerinin de÷iútirilmiú úekli (II) ve Soltis Lab. CTAB DNA ekstraksiyon protokolü (III) ve Colosi & Schall (1993)’ın DNA izolasyon yöntemleri (V) oldu÷u agaroz jel elektroforezi ve spektrofotometre ( 260 ve 280 dalga boylarında bakılarak) yöntemi ile belirlenmiútir. Bu üç yöntemden daha az zaman alan ve uygun maliyete sahip olan iki yöntem seçilerek her iki yöntem için ikiúer farklı inkübasyon süreleri uygulanarak DNA izolasyonu yapılmıútır. Farklı basamaklarda bulunan inkübasyon süreleri de÷iútirilerek, DNA miktarının yüksek, kontaminasyon oranının en düúük oldu÷u süre belirlenmiútir. Bunun sonucunda, örne÷in, CTAB çözeltisiyle kısa süreli, DNA’nın çökelmesi için kullanılan izopropanol ile uzun süreli inkübasyonun daha fazla ve daha kaliteli ürün verdi÷i elimizdeki AP-PCR primerleri yardımıyla saptanmıútır. Coyer ve ark.(1995)’nın yaptı÷ı bir çalıúmada, sıvı azotun uygulandı÷ı basamakta DNA kalitesinin ne derecede etkilendi÷i, belirlenmeye çalıúılmıútır. Bu çalıúma için üç farklı denemede toplam 12 alg örne÷i kullanılmıútır. ølk denemede örnekler, sıvı azotta ö÷ütüldükten sonra izolasyon çözeltisinde 45 dakika inkübe edilmiú, daha sonra karıúıma SDS eklenerek izolasyon aynı úekilde yürütülmüútür. Birinci denemenin ikinci grubunda ise aynı iúlem sıvı azot kullanmadan gerçekleútirilmiútir. økinci denemenin ilk grubunda, örnekler sıvı azotta ö÷ütülüp izolasyon çözeltisi ve SDS ile bir hafta inkübe edilmiútir. økinci grubunda ise aynı iúlem sıvı azot kullanılmadan tekrarlanmıútır. Son denemede ise, örnekler ö÷ütüldükten sonra santrifüj iúlemine tabi tutularak katı parçalar uzaklaútırılmıútır. øzolasyon çözeltisiyle iúlem, bu fiziksel ayrımın ardından gerçekleútirilmiútir. Sonuçlara bakıldı÷ında, ilk denemenin sıvı azot kullanılan grubundan, kullanılmayan gruba oranla % 64 daha fazla ürün elde edildi÷i görülmüútür. Ancak ikinci denemede iki grup arasındaki fark önemsenmeyecek derecede az bulunmuútur. Son denemede ise yapılan santrifüj iúleminin DNA miktarında bir etkisinin bulunmadı÷ı saptanmıútır. Coyer ve ark.(1995)’nın yapı÷ı bu çalıúmaya göre, zaman ve iú gücü gerektiren ve kullanımı zor olan sıvı azot ile ö÷ütme iúleminin, örneklerin hazırlanan izolasyon çözeltisinde 1 hafta inkübasyona bırakılması ile aynı etkiyi gösterdi÷i ileri sürülmüútür. Bunun sonucunda fazla malzemeye gereksinim olmadı÷ı için ö÷ütme iúlemi, arazi ortamında bile gerçekleútirilebilir hale getirilmiútir. Sıvı nitrojen ile ö÷ütme iúlemini tercih ediúimizin nedeni 1 haftalık inkübasyondan kaynaklanacak zaman kaybını önlemektir. Ancak laboratuvardan çok uzakta gerçekleútirilen arazi çalıúmaları sırasında, örneklerin bozulmadan saklanabilmesi için, 1 hafta boyunca izolasyon çözeltisinde ö÷ütme iúlemine bırakılması, uygun bir yöntem olacaktır. Bu çalıúmada uyguladı÷ımız yöntemde 8 saatlik inkübasyonun bulunması, gerekirse di÷er gruba izolasyon için yeterli süreyi tanıyarak, daha fazla çalıúmanın yapılabilmesine olanak sa÷lamıútır. Kullanılan kimyasalların ve cihazların laboratuvar ortamında kolaylıkla bulunabilmesi, maddi açıdan kolaylık sa÷lamıútır. øzolasyon baúına 20 mg. gibi az miktarda örne÷e gerek duyulması ve yöntemin kolay uygulanabilir olması, bu yöntemin Cystoseira mediterranea’dan DNA izolasyonu için uygun oldu÷u kanısını güçlendirmektedir. 103 SONUÇ Sonuç olarak bu çalıúmada uygulanan yöntem, C. mediterranea’nın moleküler analiz yöntemleri için, gerekli miktardaki DNA’nın kolay, hızlı ve az maliyetli bir úekilde izole edilmesine olanak vermektedir. KAYNAKLAR Colosi, Schall. 1993 CTAB miniprep DNA extraction protocol. Laboratuary Book. Coyer, J.A., Steller, D.L., Alberte, R.S. 1995. A field-competible method for extraction of fingerprint-quality DNA from Macrocystis pyrifera (Phaeophyceae). J. Phycol. 31, 177-180. Kraan, S. CTAB-PVP DNA extraction protocol. Laboratuary Book. Mayes, C., Saunders, G.W., Tan, I. H., Druehl, L.D. 1992. DNA extraction methods for kelp (Laminariales) tissue. J. Phycol 28, 712-716. Phillips, N., Smith, C.M., Morden, C.W. 2001. An effective DNA isolation protocol for brown algae. Phycological Research 49, 97-102 Saunders, G.W. 1993. Gel purification of red aldal genomic DNA: an inexpensive and rapid method for the isolation of polymerase chain reaction- friendly DNA. J. Phycol. 29, 251-254. Soltis Laboratories CTAB DNA Extraction Protocol Book. Steen, S.W. 1999. Handbook for DNA isolation, RAPD-PCR and PCR-RFLP. Botanical Garden and Museum, University of Oslo. Wattier, R.A., Prodolh, P.A., Maggs, C.A. 2000. DNA isolation protocol for red seaweed (Rhodophyta). Plant Molecular Biology Reporter 18, 275-281 104